“看清”单链DNA上每一个碱基的结构——中国科大算法辅助的针尖增强拉曼显微成像实现单碱基精度识别
- 时间:2025-10-16 15:16:49 来源:中国科大新闻网
近日,中国科学技术大学单分子科学团队的董振超研究小组在单链DNA分子结构表征领域取得重要进展,将匹配算法与贝叶斯优化结合到针尖增强拉曼光谱(TERS)分析中,实现对长链单个DNA分子的无标记结构识别与成像(图1),为高分辨无标记DNA结构表征提供了新的思路和途径。该成果于9月25日在学术期刊《JACS Au》上在线发表。
图1.机器学习辅助TERS识别长链DNA分子内单个碱基结构的艺术渲染图(AI辅助生成)
传统的高通量测序或结构表征方法常依赖标记、扩增或低温、结晶条件,可能丢失分子本征的空间取向和构型信息。TERS技术可提供无标记的高化学敏感性的亚纳米空间分辨能力,近年来作为一种无标记方法在单分子化学结构成像方面展现出巨大潜力。但此前该技术仅能对极短的单链DNA分子进行原理性演示,对长链DNA分子的精确结构分析仍面临巨大挑战,这主要是因为两个方面的原因:一是长链DNA的热稳定性和结构柔性使其实验制备和测量极其困难;二是理论上缺乏可直接计算含数万个原子的长链DNA分子拉曼光谱的模拟方法。
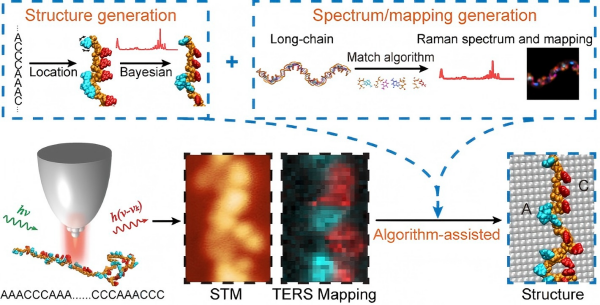
图2.算法流程图
针对这些挑战,研究团队创新性地将机器学习等算法深度融入到TERS光谱分析中,开发了一套高效的“算法-实验”协同分析策略(图2):首先开发了一种匹配算法,通过提取短链DNA中各单元(包括四种碱基、磷酸基团和戊糖)的拉曼极化率张量并利用图匹配与旋转对齐策略来匹配到长链DNA分子结构中,实现了对长链DNA的TERS光谱和空间成像图案的快速模拟。该算法巧妙绕过了传统量子化学模拟的巨大计算成本,将计算时间复杂度从O(N³)三次方依赖关系降低至O(N⟡)二次方依赖关系,如对含504个核苷酸、16130个原子的病毒DNA片段(5W7G),新方法仅需约136秒即可完成片段TERS光谱的快速计算,而传统DFT则需230余年。在此基础上,团队进一步开发了结构重建策略,以实验测量的STM图像和位点依赖的TERS光谱指纹作为初始基团位置信息的参考,利用贝叶斯算法模拟光谱,并将理论与实验光谱的相似度作为准绳优化基团取向,通过迭代最终实现对长链单根DNA模型体系单碱基级别的构型结构确定。
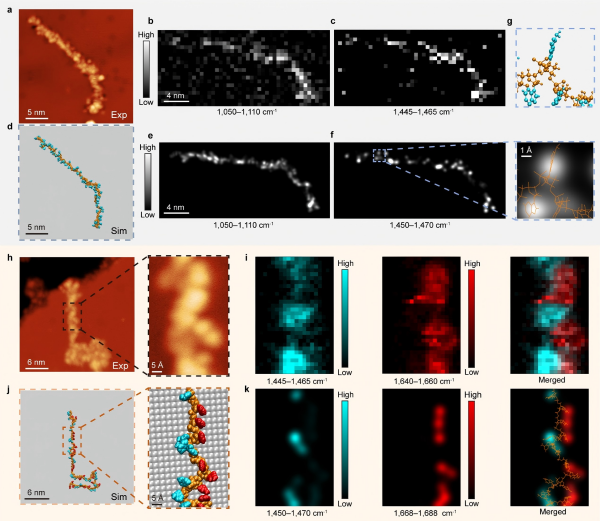
图3.A60与(AAACCC)10长链DNA分子的实验和理论结果对比
团队在采用短链DNA分子验证该算法的精确性和有效性的基础之上,以60个碱基组成的A60和(AAACCC)10这两种序列的长链DNA分子作为模型体系,对算法模型进行了进一步的实验验证。首先,在超高真空(约10-10Torr)和液氦温度(约7K)的实验条件下成功测量了两种序列分子不同位点对应的TERS光谱和高分辨成像,接着利用上述算法成功解析了对应分子的化学结构,能够在0.6nm的空间分辨率下分辨不同碱基并识别其空间取向和构型差异。通过与算法解析得到的结构对应的理论模拟TERS成像进行对比,发现二者在整体特征上有很好的对应性,特别是对于…AAACCC…序列,可以很好地区分碱基A和碱基C的不同空间分布特征(图3)。
该研究突破了传统DNA表征技术仅能获取序列信息而丢失结构信息的局限,不仅验证了TERS技术在长链DNA分子结构解析中的强大能力,也为蛋白质、糖肽复合物等其它复杂生物分子体系的立体结构解析提供了新思路。展望未来,该技术有望与纳米阵列加工及人工智能结合,发展成为一种新型的无标记生物分子测序技术,为精准药物设计和基因治疗提供技术支持。
朱路遥博士生、韩彧博士为本文共同第一作者。张尧教授、董振超教授是文章的共同通讯作者。研究工作得到了国家基金委、科技部、中国科学院、教育部、安徽省等单位的支持。
相关文章
- 中国科大开发毫秒时间分辨冷冻电镜技术,破解神经突触传递生物物理机制2025-10-17 09:00:42
- 中国科大发展“绿氢冶铁” 新技术2025-10-09 16:02:57
- 中国科大在一维极化子超晶格精密探测研究中取得重要进展2025-09-24 18:18:38
- 中国科大揭示二维笼目晶格中的电子手性可导致声子手性2025-09-22 09:02:40
- 中国科大基于频率测量法实现超高精度分子线强度比2025-09-21 08:56:04
- 中国科大构建圆偏振光学映射器件2025-09-08 10:53:03

